Die vorliegende Übersetzung wurde maschinell erstellt. Im Falle eines Konflikts oder eines Widerspruchs zwischen dieser übersetzten Fassung und der englischen Fassung (einschließlich infolge von Verzögerungen bei der Übersetzung) ist die englische Fassung maßgeblich.
Wird verwendetScriptProcessor, um den normalisierten Differenzvegetationsindex (NDVI) zu berechnen mit Sentinel-2 Satellitendaten
Die folgenden Codebeispiele zeigen Ihnen, wie Sie den normalisierten Differenzvegetationsindex eines bestimmten geografischen Gebiets mithilfe des speziell erstellten Geodatenbilds in einem Studio Classic-Notizbuch berechnen und mithilfe ScriptProcessor
Diese Demo verwendet auch eine Amazon SageMaker Studio Classic-Notebook-Instance, die den Geospatial-Kernel und den Instance-Typ verwendet. Informationen zum Erstellen einer Geospatial-Notebook-Instanz von Studio Classic finden Sie unter. Erstellen Sie ein Amazon SageMaker Studio Classic-Notizbuch mithilfe des Geodatenbilds
Sie können dieser Demo in Ihrer eigenen Notebook-Instance folgen, indem Sie die folgenden Codefragmente kopieren und einfügen:
Fragen Sie den ab Sentinel-2 Erfassung von Rasterdaten mit SearchRasterDataCollection
Mit search_raster_data_collection können Sie unterstützte Raster-Datensammlungen abfragen. In diesem Beispiel werden Daten verwendet, die abgerufen wurden aus Sentinel-2 Satelliten. Das angegebene Interessengebiet (AreaOfInterest) ist ländliches Gebiet im Norden von Iowa, und der Zeitraum (TimeRangeFilter) reicht vom 1. Januar 2022 bis 30. Dezember 2022. Um die verfügbaren Rasterdatensammlungen in Ihrem AWS-Region zu sehen, verwenden Sie list_raster_data_collections. Ein CodebeispielAPI, das dies verwendet, finden Sie ListRasterDataCollectionsim Amazon SageMaker Developer Guide.
In den folgenden Codebeispielen verwenden Sie das ARN zugehörige Sentinel-2 Erfassung von Rasterdaten,arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8.
Eine search_raster_data_collection API Anfrage erfordert zwei Parameter:
-
Sie müssen einen
ArnParameter angeben, der der Raster-Datenerfassung entspricht, die Sie abfragen möchten. -
Sie müssen auch einen
RasterDataCollectionQueryParameter angeben, der eine Python Wörterbuch.
Das folgende Codebeispiel enthält die erforderlichen Schlüssel-Wert-Paare für den Parameter RasterDataCollectionQuery, der in der Variablen search_rdc_query gespeichert wird.
search_rdc_query = { "AreaOfInterest": { "AreaOfInterestGeometry": { "PolygonGeometry": { "Coordinates": [[ [ -94.50938680498298, 43.22487436936203 ], [ -94.50938680498298, 42.843474642037194 ], [ -93.86520004156142, 42.843474642037194 ], [ -93.86520004156142, 43.22487436936203 ], [ -94.50938680498298, 43.22487436936203 ] ]] } } }, "TimeRangeFilter": {"StartTime": "2022-01-01T00:00:00Z", "EndTime": "2022-12-30T23:59:59Z"} }
Um die search_raster_data_collection Anfrage zu stellen, müssen Sie die ARN von angeben Sentinel-2 Erfassung von Raster-Daten:arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8. Sie müssen auch das zuvor definierte Python-Wörterbuch übergeben, das Abfrageparameter spezifiziert.
## Creates a SageMaker Geospatial client instance sm_geo_client= session.create_client(service_name="sagemaker-geospatial") search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query )
Die Ergebnisse API können nicht paginiert werden. Um alle von der search_raster_data_collection Operation zurückgegebenen Satellitenbilder zu sammeln, können Sie eine while Schleife implementieren. Dies prüft NextToken in der API Antwort auf:
## Holds the list of API responses from search_raster_data_collection items_list = [] while search_rdc_response1.get('NextToken') and search_rdc_response1['NextToken'] != None: items_list.extend(search_rdc_response1['Items']) search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query, NextToken=search_rdc_response1['NextToken'] )
Die API Antwort gibt eine Liste von Bändern URLs unter dem Assets Schlüssel zurück, die bestimmten Bildbändern entsprechen. Im Folgenden finden Sie eine gekürzte Version der API Antwort. Einige der Bildbänder wurden aus Gründen der Übersichtlichkeit entfernt.
{ 'Assets': { 'aot': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/AOT.tif' }, 'blue': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/B02.tif' }, 'swir22-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/B12.jp2' }, 'visual-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/TCI.jp2' }, 'wvp-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/WVP.jp2' } }, 'DateTime': datetime.datetime(2022, 12, 30, 17, 21, 52, 469000, tzinfo = tzlocal()), 'Geometry': { 'Coordinates': [ [ [-95.46676936182894, 43.32623760511659], [-94.11293433656887, 43.347431265475954], [-94.09532154452742, 42.35884880571144], [-95.42776890002203, 42.3383710796791], [-95.46676936182894, 43.32623760511659] ] ], 'Type': 'Polygon' }, 'Id': 'S2A_15TUH_20221230_0_L2A', 'Properties': { 'EoCloudCover': 62.384969, 'Platform': 'sentinel-2a' } }
Im nächsten Abschnitt erstellen Sie eine Manifestdatei mit dem 'Id' Schlüssel aus der API Antwort.
Erstellen Sie eine Eingabe-Manifestdatei mit dem Id Schlüssel aus der search_raster_data_collection API Antwort
Wenn Sie einen Verarbeitungsauftrag ausführen, müssen Sie eine Dateneingabe von Amazon S3 angeben. Der Eingabedatentyp kann entweder eine Manifestdatei sein, die dann auf die einzelnen Datendateien verweist. Sie können jeder Datei, die Sie verarbeiten möchten, auch ein Präfix hinzufügen. Das folgende Codebeispiel definiert den Ordner, in dem Ihre Manifestdateien generiert werden.
Verwenden Sie SDK für Python (Boto3), um den Standard-Bucket und die ARN Ausführungsrolle abzurufen, die Ihrer Studio Classic-Notebook-Instanz zugeordnet ist:
sm_session = sagemaker.session.Session() s3 = boto3.resource('s3') # Gets the default excution role associated with the notebook execution_role_arn = sagemaker.get_execution_role() # Gets the default bucket associated with the notebook s3_bucket = sm_session.default_bucket() # Can be replaced with any name s3_folder ="script-processor-input-manifest"
Als Nächstes erstellen Sie eine Manifestdatei. Es enthält die URLs Satellitenbilder, die Sie verarbeiten wollten, wenn Sie Ihren Verarbeitungsauftrag später in Schritt 4 ausführen.
# Format of a manifest file manifest_prefix = {} manifest_prefix['prefix'] = 's3://' + s3_bucket + '/' + s3_folder + '/' manifest = [manifest_prefix] print(manifest)
Das folgende Codebeispiel gibt das S3 zurückURI, in dem Ihre Manifestdateien erstellt werden.
[{'prefix': 's3://sagemaker-us-west-2-111122223333/script-processor-input-manifest/'}]
Alle Antwortelemente aus der search_raster_data_collection-Antwort werden nicht benötigt, um den Verarbeitungsjob auszuführen.
Der folgende Codeausschnitt entfernt die unnötigen Elemente 'Properties', 'Geometry', und 'DateTime'. Das 'Id' Schlüssel-Wert-Paar, 'Id': 'S2A_15TUH_20221230_0_L2A', enthält das Jahr und den Monat. Das folgende Codebeispiel analysiert diese Daten, um neue Schlüssel in der Python Wörterbuchdict_month_items. Die Werte sind die Assets, die von der SearchRasterDataCollection Abfrage zurückgegeben werden.
# For each response get the month and year, and then remove the metadata not related to the satelite images. dict_month_items = {} for item in items_list: # Example ID being split: 'S2A_15TUH_20221230_0_L2A' yyyymm = item['Id'].split("_")[2][:6] if yyyymm not in dict_month_items: dict_month_items[yyyymm] = [] # Removes uneeded metadata elements for this demo item.pop('Properties', None) item.pop('Geometry', None) item.pop('DateTime', None) # Appends the response from search_raster_data_collection to newly created key above dict_month_items[yyyymm].append(item)
Dieses Codebeispiel lädt das mithilfe der dict_month_items folgenden .upload_file()
## key_ is the yyyymm timestamp formatted above ## value_ is the reference to all the satellite images collected via our searchRDC query for key_, value_ in dict_month_items.items(): filename = f'manifest_{key_}.json' with open(filename, 'w') as fp: json.dump(value_, fp) s3.meta.client.upload_file(filename, s3_bucket, s3_folder + '/' + filename) manifest.append(filename) os.remove(filename)
In diesem Codebeispiel wird eine übergeordnete manifest.json Datei hochgeladen, die auf alle anderen Manifeste verweist, die auf Amazon S3 hochgeladen wurden. Es speichert auch den Pfad zu einer lokalen Variablen: s3_manifest_uri. Sie verwenden diese Variable erneut, um die Quelle der Eingabedaten anzugeben, wenn Sie den Verarbeitungsauftrag in Schritt 4 ausführen.
with open('manifest.json', 'w') as fp: json.dump(manifest, fp) s3.meta.client.upload_file('manifest.json', s3_bucket, s3_folder + '/' + 'manifest.json') os.remove('manifest.json') s3_manifest_uri = f's3://{s3_bucket}/{s3_folder}/manifest.json'
Nachdem Sie die Eingabemanifestdateien erstellt und hochgeladen haben, können Sie ein Skript schreiben, das Ihre Daten im Verarbeitungsauftrag verarbeitet. Es verarbeitet die Daten aus den Satellitenbildern, berechnet die und sendet die NDVI Ergebnisse dann an einen anderen Amazon S3 S3-Standort zurück.
Schreiben Sie ein Skript, das berechnet NDVI
Amazon SageMaker Studio Classic unterstützt die Verwendung des %%writefile Cell Magic-Befehls. Nachdem Sie eine Zelle mit diesem Befehl ausgeführt haben, wird ihr Inhalt in Ihrem lokalen Studio Classic-Verzeichnis gespeichert. Dieser Code ist spezifisch für BerechnungenNDVI. Folgendes kann jedoch nützlich sein, wenn Sie Ihr eigenes Skript für einen Verarbeitungsjob schreiben:
-
In Ihrem Verarbeitungsjob-Container müssen die lokalen Pfade innerhalb des Containers mit
/opt/ml/processing/beginnen. In diesem Beispiel werdeninput_data_path = '/opt/ml/processing/input_data/'undprocessed_data_path = '/opt/ml/processing/output_data/'auf diese Weise angegeben. -
Mit Amazon SageMaker Processing kann ein Skript, das ein Verarbeitungsauftrag ausführt, Ihre verarbeiteten Daten direkt auf Amazon S3 hochladen. Stellen Sie dazu sicher, dass die Ihrer
ScriptProcessorInstance zugeordnete Ausführungsrolle die erforderlichen Voraussetzungen für den Zugriff auf den S3-Bucket erfüllt. Sie können auch einen Ausgabeparameter angeben, wenn Sie Ihren Verarbeitungsjob ausführen. Weitere Informationen finden Sie unter Die.run()APIOperationin Amazon SageMaker Python SDK. In diesem Codebeispiel werden die Ergebnisse der Datenverarbeitung direkt auf Amazon S3 hochgeladen. -
Verwenden Sie den
volume_size_in_gbParameter, um die Größe des an Ihren Verarbeitungsauftrag EBScontainer angehängten Amazon zu verwalten. Die Standardgröße der Container ist 30 GB. Sie können optional auch die Python-Bibliothek Garbage Collectorverwenden, um den Speicher in Ihrem EBS Amazon-Container zu verwalten. Das folgende Codebeispiel lädt die Arrays in den Verarbeitungsjob-Container. Wenn sich Arrays aufbauen und den Speicher füllen, stürzt der Verarbeitungsjob ab. Um diesen Absturz zu verhindern, enthält das folgende Beispiel Befehle, mit denen die Arrays aus dem Container des Verarbeitungsjobs entfernt werden.
%%writefile compute_ndvi.py import os import pickle import sys import subprocess import json import rioxarray if __name__ == "__main__": print("Starting processing") input_data_path = '/opt/ml/processing/input_data/' input_files = [] for current_path, sub_dirs, files in os.walk(input_data_path): for file in files: if file.endswith(".json"): input_files.append(os.path.join(current_path, file)) print("Received {} input_files: {}".format(len(input_files), input_files)) items = [] for input_file in input_files: full_file_path = os.path.join(input_data_path, input_file) print(full_file_path) with open(full_file_path, 'r') as f: items.append(json.load(f)) items = [item for sub_items in items for item in sub_items] for item in items: red_uri = item["Assets"]["red"]["Href"] nir_uri = item["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) ndvi = (nir - red)/ (nir + red) file_name = 'ndvi_' + item["Id"] + '.tif' output_path = '/opt/ml/processing/output_data' output_file_path = f"{output_path}/{file_name}" ndvi.rio.to_raster(output_file_path) print("Written output:", output_file_path)
Sie haben jetzt ein Skript, das die berechnen kannNDVI. Als Nächstes können Sie eine Instanz des Verarbeitungsjobs erstellen ScriptProcessor und diesen ausführen.
Erstellen einer Instance der ScriptProcessor-Klasse
Diese Demo verwendet die ScriptProcessor.run()-Methode starten.
from sagemaker.processing import ScriptProcessor, ProcessingInput, ProcessingOutput image_uri ='081189585635.dkr.ecr.us-west-2.amazonaws.com/sagemaker-geospatial-v1-0:latest'processor = ScriptProcessor( command=['python3'], image_uri=image_uri, role=execution_role_arn, instance_count=4, instance_type='ml.m5.4xlarge', sagemaker_session=sm_session ) print('Starting processing job.')
Wenn Sie Ihren Verarbeitungsjob starten, müssen Sie ein ProcessingInput
-
Der Pfad zur Manifestdatei, die Sie in Schritt 2 erstellt haben,
s3_manifest_uri. Dies ist die Quelle der Eingabedaten für den Container. -
Der Pfad zu dem Ort, an dem die Eingabedaten im Container gespeichert werden sollen. Dieser muss mit dem Pfad übereinstimmen, den Sie in Ihrem Skript angegeben haben.
-
Verwenden Sie den Parameter
s3_data_type, um die Eingabe als"ManifestFile"zu spezifizieren.
s3_output_prefix_url = f"s3://{s3_bucket}/{s3_folder}/output" processor.run( code='compute_ndvi.py', inputs=[ ProcessingInput( source=s3_manifest_uri, destination='/opt/ml/processing/input_data/', s3_data_type="ManifestFile", s3_data_distribution_type="ShardedByS3Key" ), ], outputs=[ ProcessingOutput( source='/opt/ml/processing/output_data/', destination=s3_output_prefix_url, s3_upload_mode="Continuous" ) ] )
Im folgenden Codebeispiel wird die .describe()Methode
preprocessing_job_descriptor = processor.jobs[-1].describe() s3_output_uri = preprocessing_job_descriptor["ProcessingOutputConfig"]["Outputs"][0]["S3Output"]["S3Uri"] print(s3_output_uri)
Visualisieren Sie Ihre Ergebnisse mit matplotlib
Mit der Python-Bibliothek Matplotlib.open_rasterio() API Operation und berechnet dann die Bänder NDVI unter Verwendung der nir und der red Bildbänder aus Sentinel-2 Satellitendaten.
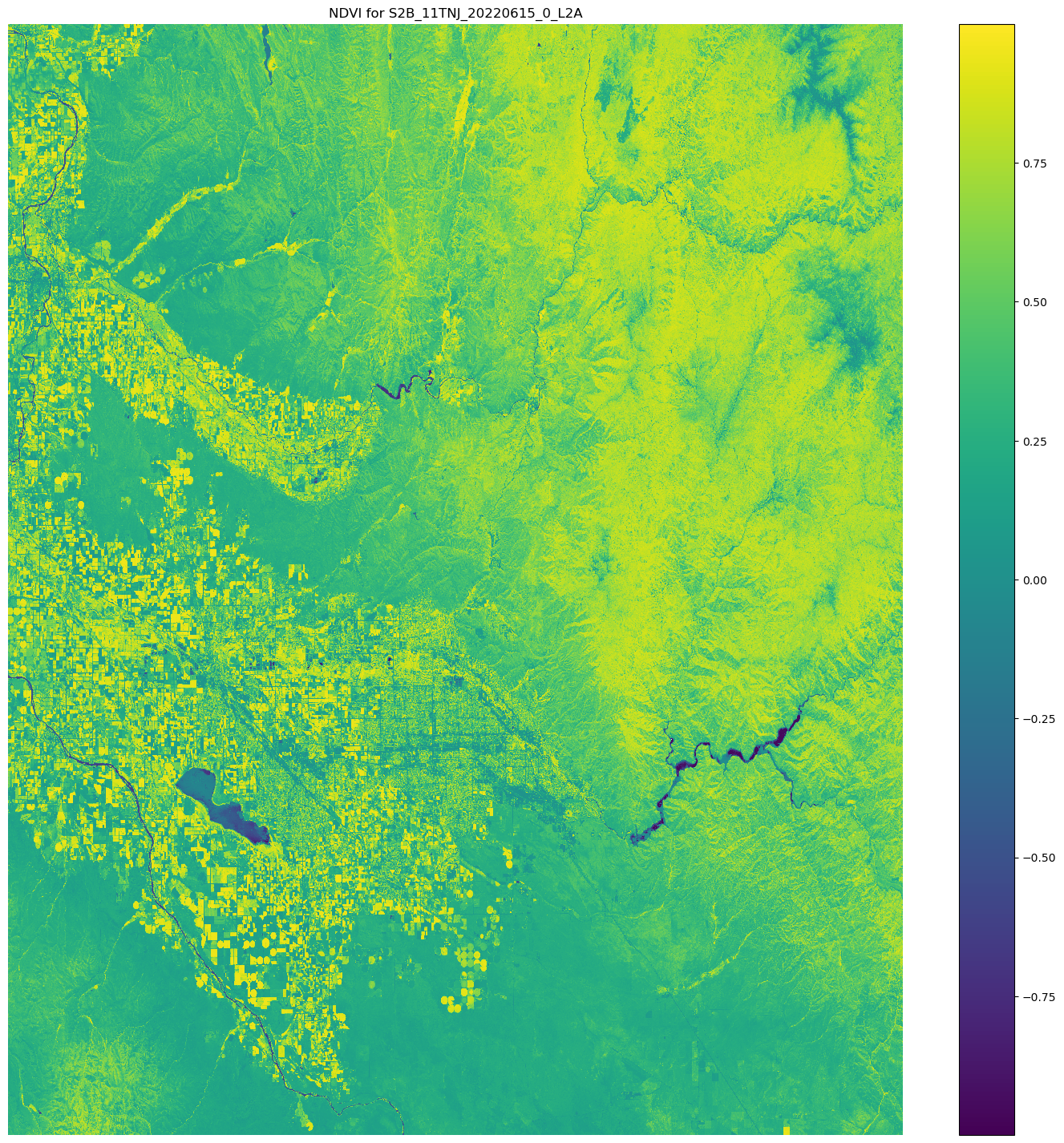
# Opens the python arrays import rioxarray red_uri = items[25]["Assets"]["red"]["Href"] nir_uri = items[25]["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) # Calculates the NDVI ndvi = (nir - red)/ (nir + red) # Common plotting library in Python import matplotlib.pyplot as plt f, ax = plt.subplots(figsize=(18, 18)) ndvi.plot(cmap='viridis', ax=ax) ax.set_title("NDVI for {}".format(items[25]["Id"])) ax.set_axis_off() plt.show()
Die Ausgabe des vorherigen Codebeispiels ist ein Satellitenbild, auf dem die NDVI Werte überlagert sind. Ein NDVI Wert nahe 1 bedeutet, dass viel Vegetation vorhanden ist, und Werte nahe 0 bedeuten, dass keine Vegetation vorhanden ist.
Damit ist die Demo der Verwendung von ScriptProcessor abgeschlossen.