Le traduzioni sono generate tramite traduzione automatica. In caso di conflitto tra il contenuto di una traduzione e la versione originale in Inglese, quest'ultima prevarrà.
Utilizzo ScriptProcessor per calcolare l'indice di vegetazione delle differenze normalizzate (NDVI) utilizzando Sentinel-2 dati satellitari
I seguenti esempi di codice mostrano come calcolare l'indice di differenza di vegetazione normalizzato di un'area geografica specifica utilizzando l'immagine geospaziale appositamente creata all'interno di un notebook Studio Classic ed eseguire un carico di lavoro su larga scala con Amazon Processing utilizzando Python. SageMaker ScriptProcessor
Questa demo utilizza anche un'istanza di notebook Amazon SageMaker Studio Classic che utilizza il kernel geospaziale e il tipo di istanza. Per informazioni su come creare un'istanza di notebook geospaziale Studio Classic, consulta. Crea un notebook Amazon SageMaker Studio Classic utilizzando l'immagine geospaziale
Puoi seguire questa demo nella tua istanza del notebook copiando e incollando i seguenti frammenti di codice:
Interroga il Sentinel-2 raccolta di dati raster utilizzando SearchRasterDataCollection
Con search_raster_data_collection puoi fare query a raccolte dei dati raster supportate. Questo esempio utilizza dati estratti da Sentinel-2 satelliti. L'area di interesse (AreaOfInterest) specificata è l'area rurale dell'Iowa settentrionale e l'intervallo temporale (TimeRangeFilter) è compreso tra il 1° gennaio 2022 e il 30 dicembre 2022. Per trovare l'elenco delle raccolte dei dati raster disponibili nella tua Regione AWS usa list_raster_data_collections. Per vedere un esempio di codice che lo utilizzaAPI, consulta ListRasterDataCollectionsla Amazon SageMaker Developer Guide.
Nei seguenti esempi di codice utilizzi il comando ARN associato a Sentinel-2 raccolta di dati raster,arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8.
Una search_raster_data_collection API richiesta richiede due parametri:
-
Devi specificare un parametro
Arnche corrisponda alla raccolta dei dati raster a cui desideri fare una query. -
È inoltre necessario specificare un
RasterDataCollectionQueryparametro, che includa un Python dizionario.
Il seguente esempio di codice contiene le coppie chiave-valore necessarie per il parametro RasterDataCollectionQuery salvato nella variabile search_rdc_query.
search_rdc_query = { "AreaOfInterest": { "AreaOfInterestGeometry": { "PolygonGeometry": { "Coordinates": [[ [ -94.50938680498298, 43.22487436936203 ], [ -94.50938680498298, 42.843474642037194 ], [ -93.86520004156142, 42.843474642037194 ], [ -93.86520004156142, 43.22487436936203 ], [ -94.50938680498298, 43.22487436936203 ] ]] } } }, "TimeRangeFilter": {"StartTime": "2022-01-01T00:00:00Z", "EndTime": "2022-12-30T23:59:59Z"} }
Per effettuare la search_raster_data_collection richiesta, è necessario specificare il ARN Sentinel-2 raccolta di dati raster:arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8. Inoltre, devi inoltrare il dizionario Python definito in precedenza, che specifica i parametri di query.
## Creates a SageMaker Geospatial client instance sm_geo_client= session.create_client(service_name="sagemaker-geospatial") search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query )
I risultati di questa operazione non API possono essere impaginati. Per raccogliere tutte le immagini satellitari restituite dall'operazione search_raster_data_collection, puoi implementare un ciclo while. Ciò verifica NextToken nella risposta: API
## Holds the list of API responses from search_raster_data_collection items_list = [] while search_rdc_response1.get('NextToken') and search_rdc_response1['NextToken'] != None: items_list.extend(search_rdc_response1['Items']) search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query, NextToken=search_rdc_response1['NextToken'] )
La API risposta restituisce un elenco di URLs sotto la Assets chiave corrispondenti a bande di immagini specifiche. Di seguito è riportata una versione troncata della risposta. API Alcune bande dell'immagine sono state rimosse per motivi di chiarezza.
{ 'Assets': { 'aot': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/AOT.tif' }, 'blue': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/B02.tif' }, 'swir22-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/B12.jp2' }, 'visual-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/TCI.jp2' }, 'wvp-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/WVP.jp2' } }, 'DateTime': datetime.datetime(2022, 12, 30, 17, 21, 52, 469000, tzinfo = tzlocal()), 'Geometry': { 'Coordinates': [ [ [-95.46676936182894, 43.32623760511659], [-94.11293433656887, 43.347431265475954], [-94.09532154452742, 42.35884880571144], [-95.42776890002203, 42.3383710796791], [-95.46676936182894, 43.32623760511659] ] ], 'Type': 'Polygon' }, 'Id': 'S2A_15TUH_20221230_0_L2A', 'Properties': { 'EoCloudCover': 62.384969, 'Platform': 'sentinel-2a' } }
Nella sezione successiva, si crea un file manifesto utilizzando la 'Id' chiave della risposta. API
Crea un file manifest di input utilizzando la Id chiave della search_raster_data_collection API risposta
Quando esegui un processo di elaborazione, devi specificare un input di dati da Amazon S3. Il tipo di dati di input può essere un file manifest che rimanda ai singoli file di dati. Inoltre puoi aggiungere un prefisso a ogni file che desideri sia elaborato. Il seguente esempio di codice definisce la cartella in cui verranno generati i file manifest.
Usa SDK per Python (Boto3) per ottenere il bucket predefinito e il ruolo ARN di esecuzione associato all'istanza del tuo notebook Studio Classic:
sm_session = sagemaker.session.Session() s3 = boto3.resource('s3') # Gets the default excution role associated with the notebook execution_role_arn = sagemaker.get_execution_role() # Gets the default bucket associated with the notebook s3_bucket = sm_session.default_bucket() # Can be replaced with any name s3_folder ="script-processor-input-manifest"
Successivamente, crea un file manifest. Conterrà le URLs immagini satellitari che desideri elaborare quando eseguirai il processo di elaborazione più avanti nel passaggio 4.
# Format of a manifest file manifest_prefix = {} manifest_prefix['prefix'] = 's3://' + s3_bucket + '/' + s3_folder + '/' manifest = [manifest_prefix] print(manifest)
Il seguente esempio di codice restituisce l'S3 URI in cui verranno creati i file manifest.
[{'prefix': 's3://sagemaker-us-west-2-111122223333/script-processor-input-manifest/'}]
Non sono necessari tutti gli elementi di risposta della risposta search_raster_data_collection per eseguire il processo di elaborazione.
Il seguente frammento di codice rimuove gli elementi non necessari 'Properties', 'Geometry' e 'DateTime'. La coppia chiave-valore'Id', 'Id': 'S2A_15TUH_20221230_0_L2A', contiene l'anno e il mese. Il seguente esempio di codice analizza tali dati per creare nuove chiavi in Python dizionario. dict_month_items I valori sono gli asset restituiti dalla query SearchRasterDataCollection.
# For each response get the month and year, and then remove the metadata not related to the satelite images. dict_month_items = {} for item in items_list: # Example ID being split: 'S2A_15TUH_20221230_0_L2A' yyyymm = item['Id'].split("_")[2][:6] if yyyymm not in dict_month_items: dict_month_items[yyyymm] = [] # Removes uneeded metadata elements for this demo item.pop('Properties', None) item.pop('Geometry', None) item.pop('DateTime', None) # Appends the response from search_raster_data_collection to newly created key above dict_month_items[yyyymm].append(item)
Questo esempio di codice carica su Amazon S3 come JSON oggetto utilizzando .upload_file()dict_month_items
## key_ is the yyyymm timestamp formatted above ## value_ is the reference to all the satellite images collected via our searchRDC query for key_, value_ in dict_month_items.items(): filename = f'manifest_{key_}.json' with open(filename, 'w') as fp: json.dump(value_, fp) s3.meta.client.upload_file(filename, s3_bucket, s3_folder + '/' + filename) manifest.append(filename) os.remove(filename)
Questo esempio di codice carica un file parent manifest.json che rimanda a tutti gli altri manifest caricati su Amazon S3. Salva anche il percorso di una variabile locale: s3_manifest_uri. Utilizzerai nuovamente quella variabile per specificare l’origine dei dati di input quando eseguirai il processo di elaborazione nella fase 4.
with open('manifest.json', 'w') as fp: json.dump(manifest, fp) s3.meta.client.upload_file('manifest.json', s3_bucket, s3_folder + '/' + 'manifest.json') os.remove('manifest.json') s3_manifest_uri = f's3://{s3_bucket}/{s3_folder}/manifest.json'
Ora che hai creato e caricato i file manifest di input, puoi scrivere uno script che elabori i tuoi dati durante il processo di elaborazione. Elabora i dati delle immagini satellitari, calcola e quindi restituisce i risultati in una diversa posizione di Amazon S3. NDVI
Scrivi uno script che calcoli il NDVI
Amazon SageMaker Studio Classic supporta l'uso del comando %%writefile cell magic. Dopo aver eseguito una cella con questo comando, il suo contenuto verrà salvato nella directory locale di Studio Classic. Si tratta di un codice specifico per il calcolo. NDVI Tuttavia, quanto segue può essere utile quando scrivi uno script personalizzato per un processo di elaborazione:
-
I percorsi locali all'interno di un container di processi di elaborazione devono iniziare con
/opt/ml/processing/. In questo esempio,input_data_path = '/opt/ml/processing/input_data/'eprocessed_data_path = '/opt/ml/processing/output_data/'sono specificati in questo modo. -
Con Amazon SageMaker Processing, uno script eseguito da un processo di elaborazione può caricare i dati elaborati direttamente su Amazon S3. A tal fine, assicurati che il ruolo di esecuzione associato alla tua istanza
ScriptProcessorabbia i requisiti necessari per accedere al bucket S3. Puoi anche specificare un parametro di output quando esegui il processo di elaborazione. Per ulteriori informazioni, consulta il.run()APIfunzionamentoin Amazon SageMaker Python SDK. In questo esempio di codice, i risultati dell'elaborazione di dati vengono caricati direttamente in Amazon S3. -
Per gestire le dimensioni dell'Amazon EBScontainer collegato al processo di elaborazione, utilizza il
volume_size_in_gbparametro. La dimensione predefinita dei container è 30 GB. Facoltativamente, puoi anche utilizzare la libreria Python Garbage Collector pergestire lo storage nel tuo contenitore Amazon. EBS Il seguente esempio di codice carica gli array nel container del processo di elaborazione. Quando gli array si accumulano e riempiono la memoria, il processo di elaborazione si arresta in modo anomalo. Per evitare questo arresto anomalo, l'esempio seguente contiene comandi che rimuovono gli array dal container del processo di elaborazione.
%%writefile compute_ndvi.py import os import pickle import sys import subprocess import json import rioxarray if __name__ == "__main__": print("Starting processing") input_data_path = '/opt/ml/processing/input_data/' input_files = [] for current_path, sub_dirs, files in os.walk(input_data_path): for file in files: if file.endswith(".json"): input_files.append(os.path.join(current_path, file)) print("Received {} input_files: {}".format(len(input_files), input_files)) items = [] for input_file in input_files: full_file_path = os.path.join(input_data_path, input_file) print(full_file_path) with open(full_file_path, 'r') as f: items.append(json.load(f)) items = [item for sub_items in items for item in sub_items] for item in items: red_uri = item["Assets"]["red"]["Href"] nir_uri = item["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) ndvi = (nir - red)/ (nir + red) file_name = 'ndvi_' + item["Id"] + '.tif' output_path = '/opt/ml/processing/output_data' output_file_path = f"{output_path}/{file_name}" ndvi.rio.to_raster(output_file_path) print("Written output:", output_file_path)
Ora hai uno script in grado di calcolare il. NDVI Successivamente, è possibile creare un'istanza di ScriptProcessor ed eseguire il processo Processing.
Creazione di un'istanza della classe ScriptProcessor
Questa demo utilizza la ScriptProcessor.run().
from sagemaker.processing import ScriptProcessor, ProcessingInput, ProcessingOutput image_uri ='081189585635.dkr.ecr.us-west-2.amazonaws.com/sagemaker-geospatial-v1-0:latest'processor = ScriptProcessor( command=['python3'], image_uri=image_uri, role=execution_role_arn, instance_count=4, instance_type='ml.m5.4xlarge', sagemaker_session=sm_session ) print('Starting processing job.')
Quando avvii il processo di elaborazione, devi specificare un oggetto ProcessingInput
-
Il percorso del file manifest creato nella fase 2,
s3_manifest_uri. Questa è l’origine dei dati di input per il container. -
Il percorso in cui desideri che i dati di input vengano salvati nel container. Deve corrispondere al percorso specificato nello script.
-
Utilizza il parametro
s3_data_typeper specificare l’input come"ManifestFile".
s3_output_prefix_url = f"s3://{s3_bucket}/{s3_folder}/output" processor.run( code='compute_ndvi.py', inputs=[ ProcessingInput( source=s3_manifest_uri, destination='/opt/ml/processing/input_data/', s3_data_type="ManifestFile", s3_data_distribution_type="ShardedByS3Key" ), ], outputs=[ ProcessingOutput( source='/opt/ml/processing/output_data/', destination=s3_output_prefix_url, s3_upload_mode="Continuous" ) ] )
Il seguente esempio di codice utilizza il metodo .describe()
preprocessing_job_descriptor = processor.jobs[-1].describe() s3_output_uri = preprocessing_job_descriptor["ProcessingOutputConfig"]["Outputs"][0]["S3Output"]["S3Uri"] print(s3_output_uri)
Visualizzazione dei risultati utilizzando matplotlib
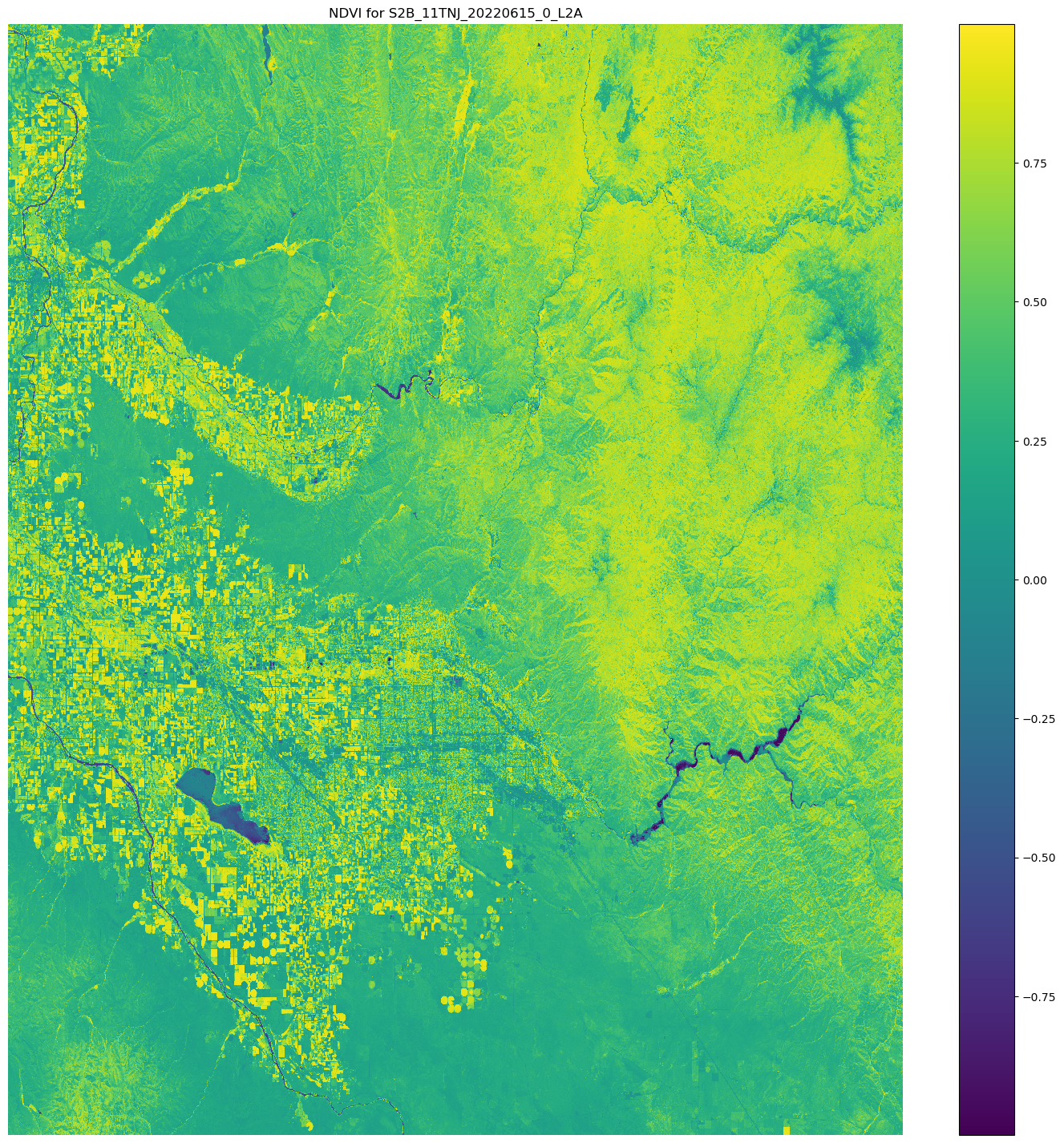
La libreria Matplotlib.open_rasterio()APIoperazione, quindi calcola l'NDVIutilizzo delle bande di red immagini nir e delle Sentinel-2 dati satellitari.
# Opens the python arrays import rioxarray red_uri = items[25]["Assets"]["red"]["Href"] nir_uri = items[25]["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) # Calculates the NDVI ndvi = (nir - red)/ (nir + red) # Common plotting library in Python import matplotlib.pyplot as plt f, ax = plt.subplots(figsize=(18, 18)) ndvi.plot(cmap='viridis', ax=ax) ax.set_title("NDVI for {}".format(items[25]["Id"])) ax.set_axis_off() plt.show()
L'output dell'esempio di codice precedente è un'immagine satellitare con i NDVI valori sovrapposti. Un NDVI valore vicino a 1 indica che è presente molta vegetazione, mentre valori vicini a 0 indicano che non è presente vegetazione.
Questo completa la demo sull’utilizzo di ScriptProcessor.