Las traducciones son generadas a través de traducción automática. En caso de conflicto entre la traducción y la version original de inglés, prevalecerá la version en inglés.
Se utiliza ScriptProcessor para calcular la diferencia normalizada del índice de vegetación (NDVI) mediante Sentinel-2 datos de satélite
En los siguientes ejemplos de código, se muestra cómo calcular el índice de vegetación diferencial normalizado de un área geográfica específica mediante la imagen geoespacial diseñada específicamente en un bloc de notas de Studio Classic y cómo ejecutar una carga de ScriptProcessor
Esta demostración también usa una instancia de bloc de notas Amazon SageMaker Studio Classic que usa el kernel geoespacial y el tipo de instancia. Para obtener información sobre cómo crear una instancia de bloc de notas geoespacial de Studio Classic, consulte. Cree un bloc de notas Amazon SageMaker Studio Classic con la imagen geoespacial
Puede seguir esta demostración en su propia instancia de cuaderno copiando y pegando los siguientes fragmentos de código:
Consulta el Sentinel-2 recopilación de datos ráster mediante SearchRasterDataCollection
Con search_raster_data_collection puede consultar las colecciones de datos ráster compatibles. En este ejemplo se utilizan datos extraídos de Sentinel-2 satélites. El área de interés especificada (AreaOfInterest) es una zona rural del norte de Iowa y el intervalo de tiempo (TimeRangeFilter) va del 1 de enero de 2022 al 30 de diciembre de 2022. Para ver las recopilaciones de datos ráster disponibles en su Región de AWS , use list_raster_data_collections. Para ver un ejemplo de código con estoAPI, consulta ListRasterDataCollectionsla Guía para SageMaker desarrolladores de Amazon.
En los siguientes ejemplos de código, utiliza el código ARN asociado a Sentinel-2 recopilación de datos ráster,arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8.
Una search_raster_data_collection API solicitud requiere dos parámetros:
-
Debe especificar un parámetro
Arnque corresponda a la recopilación de datos ráster que desea consultar. -
También debe especificar un
RasterDataCollectionQueryparámetro, que incluya un Python diccionario.
El siguiente ejemplo de código contiene los pares clave-valor necesarios para el parámetro RasterDataCollectionQuery guardado en la variable search_rdc_query.
search_rdc_query = { "AreaOfInterest": { "AreaOfInterestGeometry": { "PolygonGeometry": { "Coordinates": [[ [ -94.50938680498298, 43.22487436936203 ], [ -94.50938680498298, 42.843474642037194 ], [ -93.86520004156142, 42.843474642037194 ], [ -93.86520004156142, 43.22487436936203 ], [ -94.50938680498298, 43.22487436936203 ] ]] } } }, "TimeRangeFilter": {"StartTime": "2022-01-01T00:00:00Z", "EndTime": "2022-12-30T23:59:59Z"} }
Para realizar la search_raster_data_collection solicitud, debe especificar ARN el Sentinel-2 recopilación de datos ráster:arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8. También debe pasar el diccionario de Python que se definió anteriormente, que especifica los parámetros de consulta.
## Creates a SageMaker Geospatial client instance sm_geo_client= session.create_client(service_name="sagemaker-geospatial") search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query )
Los resultados de esto no API se pueden paginar. Para recopilar todas las imágenes de satélite devueltas por la operación search_raster_data_collection, puede implementar un bucle de tipo while. Esto comprueba NextToken en la API respuesta:
## Holds the list of API responses from search_raster_data_collection items_list = [] while search_rdc_response1.get('NextToken') and search_rdc_response1['NextToken'] != None: items_list.extend(search_rdc_response1['Items']) search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query, NextToken=search_rdc_response1['NextToken'] )
La API respuesta devuelve una lista de URLs debajo de la Assets clave correspondiente a bandas de imagen específicas. La siguiente es una versión truncada de la API respuesta. Hemos quitado algunas de las bandas de la imagen para ofrecer una mayor claridad.
{ 'Assets': { 'aot': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/AOT.tif' }, 'blue': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/B02.tif' }, 'swir22-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/B12.jp2' }, 'visual-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/TCI.jp2' }, 'wvp-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/WVP.jp2' } }, 'DateTime': datetime.datetime(2022, 12, 30, 17, 21, 52, 469000, tzinfo = tzlocal()), 'Geometry': { 'Coordinates': [ [ [-95.46676936182894, 43.32623760511659], [-94.11293433656887, 43.347431265475954], [-94.09532154452742, 42.35884880571144], [-95.42776890002203, 42.3383710796791], [-95.46676936182894, 43.32623760511659] ] ], 'Type': 'Polygon' }, 'Id': 'S2A_15TUH_20221230_0_L2A', 'Properties': { 'EoCloudCover': 62.384969, 'Platform': 'sentinel-2a' } }
En la siguiente sección, crearás un archivo de manifiesto con la 'Id' clave de la API respuesta.
Crea un archivo de manifiesto de entrada con la Id clave de la search_raster_data_collection API respuesta
Al ejecutar un trabajo de procesamiento, debe especificar una entrada de datos de Amazon S3. El tipo de datos de entrada puede ser un archivo de manifiesto, que luego apunta a los archivos de datos individuales. También puede agregar un prefijo a cada archivo que desee procesar. El siguiente ejemplo de código define la carpeta en la que se generarán los archivos de manifiesto.
Utilízalo SDK para Python (Boto3) para obtener el bucket predeterminado y el rol ARN de ejecución que está asociado a tu instancia de bloc de notas de Studio Classic:
sm_session = sagemaker.session.Session() s3 = boto3.resource('s3') # Gets the default excution role associated with the notebook execution_role_arn = sagemaker.get_execution_role() # Gets the default bucket associated with the notebook s3_bucket = sm_session.default_bucket() # Can be replaced with any name s3_folder ="script-processor-input-manifest"
A continuación, creará un archivo de manifiesto. Contendrá las imágenes URLs de satélite que desee procesar cuando ejecute el trabajo de procesamiento más adelante en el paso 4.
# Format of a manifest file manifest_prefix = {} manifest_prefix['prefix'] = 's3://' + s3_bucket + '/' + s3_folder + '/' manifest = [manifest_prefix] print(manifest)
El siguiente ejemplo de código devuelve el S3 en el URI que se crearán los archivos de manifiesto.
[{'prefix': 's3://sagemaker-us-west-2-111122223333/script-processor-input-manifest/'}]
No se necesitan todos los elementos de respuesta de la respuesta search_raster_data_collection para ejecutar el trabajo de procesamiento.
El siguiente fragmento de código elimina los elementos innecesarios 'Properties', 'Geometry' y 'DateTime'. El par clave-valor 'Id', 'Id': 'S2A_15TUH_20221230_0_L2A', contiene el año y el mes. El siguiente ejemplo de código analiza esos datos para crear nuevas claves en el Python diccionariodict_month_items. Los valores son los activos que se devuelven desde la consulta SearchRasterDataCollection.
# For each response get the month and year, and then remove the metadata not related to the satelite images. dict_month_items = {} for item in items_list: # Example ID being split: 'S2A_15TUH_20221230_0_L2A' yyyymm = item['Id'].split("_")[2][:6] if yyyymm not in dict_month_items: dict_month_items[yyyymm] = [] # Removes uneeded metadata elements for this demo item.pop('Properties', None) item.pop('Geometry', None) item.pop('DateTime', None) # Appends the response from search_raster_data_collection to newly created key above dict_month_items[yyyymm].append(item)
En este ejemplo de código, se carga el dict_month_items JSON objeto en Amazon S3 mediante la .upload_file()
## key_ is the yyyymm timestamp formatted above ## value_ is the reference to all the satellite images collected via our searchRDC query for key_, value_ in dict_month_items.items(): filename = f'manifest_{key_}.json' with open(filename, 'w') as fp: json.dump(value_, fp) s3.meta.client.upload_file(filename, s3_bucket, s3_folder + '/' + filename) manifest.append(filename) os.remove(filename)
En este ejemplo de código se carga un archivo manifest.json principal que apunta a todos los demás manifiestos subidos a Amazon S3. También guarda la ruta a una variable local: s3_manifest_uri. Volverá a utilizar esa variable para especificar la fuente de los datos de entrada cuando ejecute el trabajo de procesamiento en el paso 4.
with open('manifest.json', 'w') as fp: json.dump(manifest, fp) s3.meta.client.upload_file('manifest.json', s3_bucket, s3_folder + '/' + 'manifest.json') os.remove('manifest.json') s3_manifest_uri = f's3://{s3_bucket}/{s3_folder}/manifest.json'
Ahora que ha creado los archivos de manifiesto de entrada y los ha cargado, puede escribir un script que procese los datos en el trabajo de procesamiento. Procesa los datos de las imágenes de satélite, calcula yNDVI, a continuación, devuelve los resultados a una ubicación diferente de Amazon S3.
Escriba un script que calcule la NDVI
Amazon SageMaker Studio Classic admite el uso del comando %%writefile cell magic. Tras ejecutar una celda con este comando, su contenido se guardará en el directorio local de Studio Classic. Se trata de un código específico para el cálculoNDVI. Sin embargo, lo que explicamos a continuación puede resultar útil a la hora de escribir su propio script para un trabajo de procesamiento:
-
En el contenedor de tareas de procesamiento, las rutas locales dentro del contenedor deben empezar por
/opt/ml/processing/. En este ejemplo,input_data_path = '/opt/ml/processing/input_data/'yprocessed_data_path = '/opt/ml/processing/output_data/'se especifican de esa manera. -
Con Amazon SageMaker Processing, un script que ejecute un trabajo de procesamiento puede cargar los datos procesados directamente en Amazon S3. Para ello, asegúrese de que la función de ejecución asociada a la instancia
ScriptProcessorcumpla los requisitos necesarios para acceder al bucket de S3. También puede especificar un parámetro de salida al ejecutar el trabajo de procesamiento. Para obtener más información, consulte la.run()APIoperaciónen Amazon SageMaker Python SDK. En este ejemplo de código, los resultados del procesamiento de datos se cargan directamente en Amazon S3. -
Para gestionar el tamaño del Amazon EBScontainer adjunto a tu trabajo de procesamiento, usa el
volume_size_in_gbparámetro. El tamaño predeterminado de los contenedores es de 30 GB. Opcionalmente, también puedes usar la biblioteca de Python Garbage Collectorpara administrar el almacenamiento en tu EBS contenedor de Amazon. El siguiente ejemplo de código carga las matrices en el contenedor de tareas de procesamiento. Cuando las matrices se acumulan y llenan la memoria, el trabajo de procesamiento se bloquea. Para evitar este bloqueo, el siguiente ejemplo contiene comandos que eliminan las matrices del contenedor del trabajo de procesamiento.
%%writefile compute_ndvi.py import os import pickle import sys import subprocess import json import rioxarray if __name__ == "__main__": print("Starting processing") input_data_path = '/opt/ml/processing/input_data/' input_files = [] for current_path, sub_dirs, files in os.walk(input_data_path): for file in files: if file.endswith(".json"): input_files.append(os.path.join(current_path, file)) print("Received {} input_files: {}".format(len(input_files), input_files)) items = [] for input_file in input_files: full_file_path = os.path.join(input_data_path, input_file) print(full_file_path) with open(full_file_path, 'r') as f: items.append(json.load(f)) items = [item for sub_items in items for item in sub_items] for item in items: red_uri = item["Assets"]["red"]["Href"] nir_uri = item["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) ndvi = (nir - red)/ (nir + red) file_name = 'ndvi_' + item["Id"] + '.tif' output_path = '/opt/ml/processing/output_data' output_file_path = f"{output_path}/{file_name}" ndvi.rio.to_raster(output_file_path) print("Written output:", output_file_path)
Ahora tiene un script que puede calcular elNDVI. A continuación, puede crear una instancia ScriptProcessor y ejecutar su trabajo de procesamiento.
Creación de una instancia de la clase ScriptProcessor
Esta demostración usa la ScriptProcessor.run().
from sagemaker.processing import ScriptProcessor, ProcessingInput, ProcessingOutput image_uri ='081189585635.dkr.ecr.us-west-2.amazonaws.com/sagemaker-geospatial-v1-0:latest'processor = ScriptProcessor( command=['python3'], image_uri=image_uri, role=execution_role_arn, instance_count=4, instance_type='ml.m5.4xlarge', sagemaker_session=sm_session ) print('Starting processing job.')
Al iniciar el trabajo de procesamiento, debe especificar un objeto ProcessingInput
-
La ruta al archivo de manifiesto que creó en el paso 2,
s3_manifest_uri. Este es el origen de los datos de entrada al contenedor. -
La ruta en la que desea que se guarden los datos de entrada en el contenedor. Debe coincidir con la ruta que especificó en el script.
-
Use el parámetro
s3_data_typepara especificar la entrada como"ManifestFile".
s3_output_prefix_url = f"s3://{s3_bucket}/{s3_folder}/output" processor.run( code='compute_ndvi.py', inputs=[ ProcessingInput( source=s3_manifest_uri, destination='/opt/ml/processing/input_data/', s3_data_type="ManifestFile", s3_data_distribution_type="ShardedByS3Key" ), ], outputs=[ ProcessingOutput( source='/opt/ml/processing/output_data/', destination=s3_output_prefix_url, s3_upload_mode="Continuous" ) ] )
El siguiente ejemplo de código utiliza el método .describe()
preprocessing_job_descriptor = processor.jobs[-1].describe() s3_output_uri = preprocessing_job_descriptor["ProcessingOutputConfig"]["Outputs"][0]["S3Output"]["S3Uri"] print(s3_output_uri)
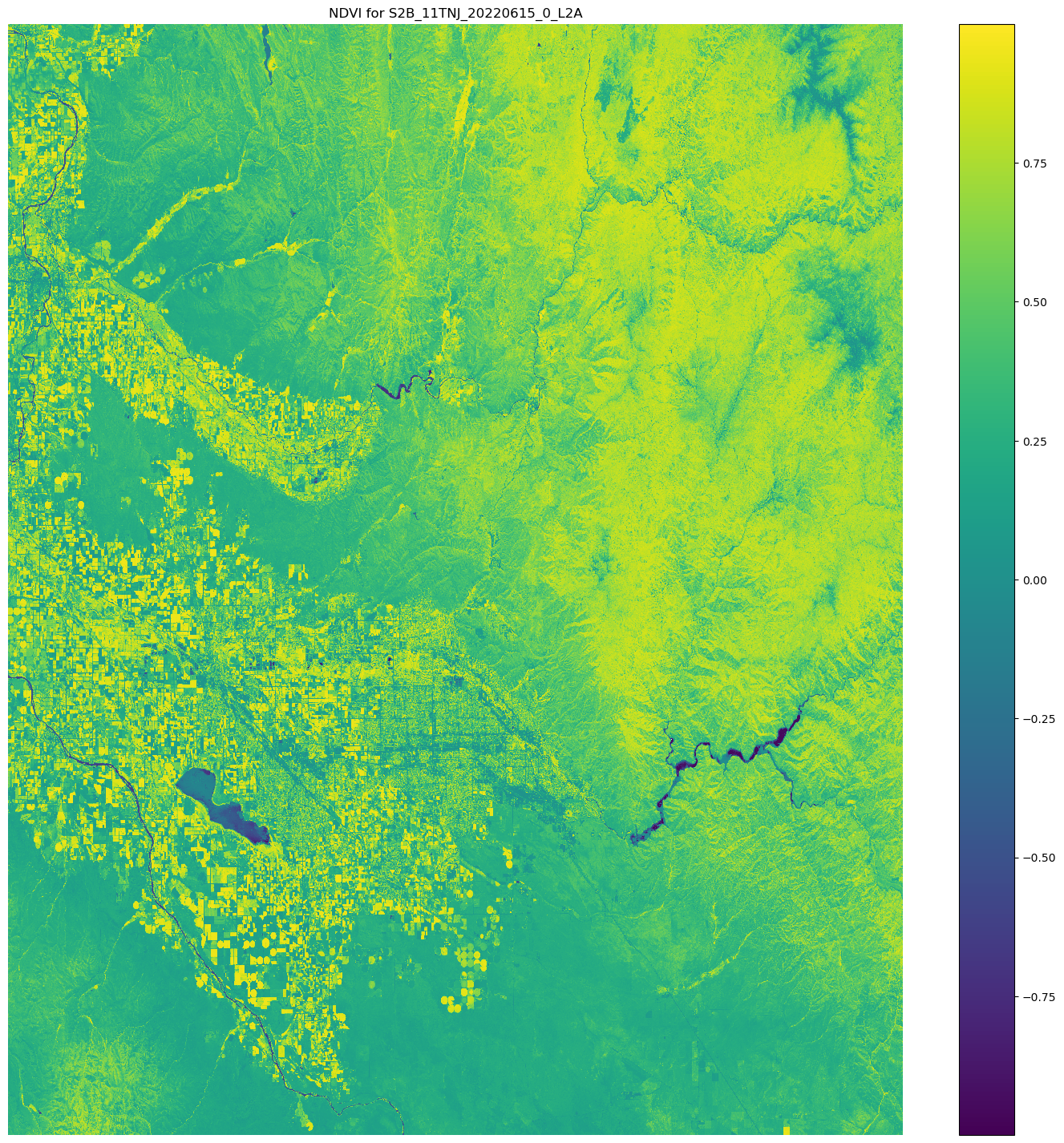
Visualización de los resultados mediante matplotlib
Con la biblioteca de Python Matplotlib.open_rasterio() API operación y, a continuación, se calculan las bandas NDVI mediante las bandas de red imágenes nir y procedentes del Sentinel-2 datos de satélite.
# Opens the python arrays import rioxarray red_uri = items[25]["Assets"]["red"]["Href"] nir_uri = items[25]["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) # Calculates the NDVI ndvi = (nir - red)/ (nir + red) # Common plotting library in Python import matplotlib.pyplot as plt f, ax = plt.subplots(figsize=(18, 18)) ndvi.plot(cmap='viridis', ax=ax) ax.set_title("NDVI for {}".format(items[25]["Id"])) ax.set_axis_off() plt.show()
El resultado del ejemplo de código anterior es una imagen de satélite con los NDVI valores superpuestos. Un NDVI valor cercano a 1 indica que hay mucha vegetación y los valores cercanos a 0 indican que no hay vegetación.
Esto completa la demostración de uso de ScriptProcessor.