Les traductions sont fournies par des outils de traduction automatique. En cas de conflit entre le contenu d'une traduction et celui de la version originale en anglais, la version anglaise prévaudra.
Les exemples de code suivants vous montrent comment calculer l'indice de végétation différentiel normalisé d'une zone géographique spécifique à l'aide de l'image géospatiale spécialement conçue dans un bloc-notes Studio Classic et comment exécuter une charge de travail à grande échelle avec Amazon SageMaker Processing à l'aide ScriptProcessor
Cette démonstration utilise également une instance de bloc-notes Amazon SageMaker Studio Classic qui utilise le noyau géospatial et le type d'instance. Pour savoir comment créer une instance de bloc-notes géospatial Studio Classic, consultezCréation d'un bloc-notes Amazon SageMaker Studio Classic à l'aide de l'image géospatiale.
Vous pouvez suivre cette démonstration dans votre propre instance de bloc-notes en copiant et en collant les extraits de code suivants :
Interrogez le Sentinel-2 collecte de données raster à l'aide de SearchRasterDataCollection
search_raster_data_collectionVous pouvez ainsi interroger les collections de données raster prises en charge. Cet exemple utilise des données extraites de Sentinel-2 satellites. La zone d'intérêt (AreaOfInterest) spécifiée est la zone rurale du nord de l'Iowa, et la période (TimeRangeFilter) va du 1er janvier 2022 au 30 décembre 2022. Pour voir les collections de données raster disponibles que vous Région AWS utilisezlist_raster_data_collections. Pour voir un exemple de code utilisant cette API, consultez ListRasterDataCollectionsle manuel Amazon SageMaker AI Developer Guide.
Dans les exemples de code suivants, vous utilisez l'ARN associé à Sentinel-2 collecte de données matricielles,arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8.
Une demande d'search_raster_data_collectionAPI nécessite deux paramètres :
-
Vous devez spécifier un
Arnparamètre correspondant à la collection de données raster que vous souhaitez interroger. -
Vous devez également spécifier un
RasterDataCollectionQueryparamètre, qui prend en compte un Python dictionnaire.
L'exemple de code suivant contient les paires clé-valeur nécessaires pour le RasterDataCollectionQuery paramètre enregistré dans la search_rdc_query variable.
search_rdc_query = {
"AreaOfInterest": {
"AreaOfInterestGeometry": {
"PolygonGeometry": {
"Coordinates": [[
[
-94.50938680498298,
43.22487436936203
],
[
-94.50938680498298,
42.843474642037194
],
[
-93.86520004156142,
42.843474642037194
],
[
-93.86520004156142,
43.22487436936203
],
[
-94.50938680498298,
43.22487436936203
]
]]
}
}
},
"TimeRangeFilter": {"StartTime": "2022-01-01T00:00:00Z", "EndTime": "2022-12-30T23:59:59Z"}
}Pour effectuer la search_raster_data_collection demande, vous devez spécifier l'ARN du Sentinel-2 collecte de données matricielles :arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8. Vous devez également transmettre le dictionnaire Python défini précédemment, qui spécifie les paramètres de requête.
## Creates a SageMaker Geospatial client instance
sm_geo_client= session.create_client(service_name="sagemaker-geospatial")
search_rdc_response1 = sm_geo_client.search_raster_data_collection(
Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8',
RasterDataCollectionQuery=search_rdc_query
)Les résultats de cette API ne peuvent pas être paginés. Pour collecter toutes les images satellites renvoyées par l'search_raster_data_collectionopération, vous pouvez implémenter une while boucle. Cela vérifie NextToken dans la réponse de l'API :
## Holds the list of API responses from search_raster_data_collection
items_list = []
while search_rdc_response1.get('NextToken') and search_rdc_response1['NextToken'] != None:
items_list.extend(search_rdc_response1['Items'])
search_rdc_response1 = sm_geo_client.search_raster_data_collection(
Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8',
RasterDataCollectionQuery=search_rdc_query,
NextToken=search_rdc_response1['NextToken']
)La réponse de l'API renvoie une liste de URLs Assets sous-touches correspondant à des bandes d'images spécifiques. Voici une version tronquée de la réponse de l'API. Certaines bandes d'image ont été supprimées pour des raisons de clarté.
{
'Assets': {
'aot': {
'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/AOT.tif'
},
'blue': {
'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/B02.tif'
},
'swir22-jp2': {
'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/B12.jp2'
},
'visual-jp2': {
'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/TCI.jp2'
},
'wvp-jp2': {
'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/WVP.jp2'
}
},
'DateTime': datetime.datetime(2022, 12, 30, 17, 21, 52, 469000, tzinfo = tzlocal()),
'Geometry': {
'Coordinates': [
[
[-95.46676936182894, 43.32623760511659],
[-94.11293433656887, 43.347431265475954],
[-94.09532154452742, 42.35884880571144],
[-95.42776890002203, 42.3383710796791],
[-95.46676936182894, 43.32623760511659]
]
],
'Type': 'Polygon'
},
'Id': 'S2A_15TUH_20221230_0_L2A',
'Properties': {
'EoCloudCover': 62.384969,
'Platform': 'sentinel-2a'
}
}Dans la section suivante, vous allez créer un fichier manifeste à l'aide de la 'Id' clé de la réponse de l'API.
Créez un fichier manifeste d'entrée à l'aide de la Id clé de la réponse de l'search_raster_data_collectionAPI
Lorsque vous exécutez une tâche de traitement, vous devez spécifier une entrée de données provenant d'Amazon S3. Le type de données d'entrée peut être un fichier manifeste, qui pointe ensuite vers les fichiers de données individuels. Vous pouvez également ajouter un préfixe à chaque fichier que vous souhaitez traiter. L'exemple de code suivant définit le dossier dans lequel vos fichiers manifestes seront générés.
Utilisez le SDK pour Python (Boto3) pour obtenir le bucket par défaut et l'ARN du rôle d'exécution associé à votre instance de bloc-notes Studio Classic :
sm_session = sagemaker.session.Session()
s3 = boto3.resource('s3')
# Gets the default excution role associated with the notebook
execution_role_arn = sagemaker.get_execution_role()
# Gets the default bucket associated with the notebook
s3_bucket = sm_session.default_bucket()
# Can be replaced with any name
s3_folder = "script-processor-input-manifest"Ensuite, vous créez un fichier manifeste. Il contiendra les URLs images satellites que vous souhaitez traiter lorsque vous exécuterez votre tâche de traitement ultérieurement à l'étape 4.
# Format of a manifest file
manifest_prefix = {}
manifest_prefix['prefix'] = 's3://' + s3_bucket + '/' + s3_folder + '/'
manifest = [manifest_prefix]
print(manifest)L'exemple de code suivant renvoie l'URI S3 dans lequel vos fichiers manifestes seront créés.
[{'prefix': 's3://sagemaker-us-west-2-111122223333/script-processor-input-manifest/'}]Tous les éléments de réponse de la réponse search_raster_data_collection ne sont pas nécessaires pour exécuter la tâche de traitement.
L'extrait de code suivant supprime les éléments inutiles 'Properties''Geometry', et. 'DateTime' La paire 'Id' clé-valeur contient 'Id': 'S2A_15TUH_20221230_0_L2A' l'année et le mois. L'exemple de code suivant analyse ces données pour créer de nouvelles clés dans Python dictionnairedict_month_items. Les valeurs sont les actifs renvoyés par la SearchRasterDataCollection requête.
# For each response get the month and year, and then remove the metadata not related to the satelite images.
dict_month_items = {}
for item in items_list:
# Example ID being split: 'S2A_15TUH_20221230_0_L2A'
yyyymm = item['Id'].split("_")[2][:6]
if yyyymm not in dict_month_items:
dict_month_items[yyyymm] = []
# Removes uneeded metadata elements for this demo
item.pop('Properties', None)
item.pop('Geometry', None)
item.pop('DateTime', None)
# Appends the response from search_raster_data_collection to newly created key above
dict_month_items[yyyymm].append(item)Cet exemple de code télécharge le dict_month_items vers Amazon S3 en tant qu'objet JSON à l'aide de l'opération .upload_file()
## key_ is the yyyymm timestamp formatted above
## value_ is the reference to all the satellite images collected via our searchRDC query
for key_, value_ in dict_month_items.items():
filename = f'manifest_{key_}.json'
with open(filename, 'w') as fp:
json.dump(value_, fp)
s3.meta.client.upload_file(filename, s3_bucket, s3_folder + '/' + filename)
manifest.append(filename)
os.remove(filename)Cet exemple de code télécharge un manifest.json fichier parent qui pointe vers tous les autres manifestes chargés sur Amazon S3. Il enregistre également le chemin d'une variable locale :s3_manifest_uri. Vous utiliserez à nouveau cette variable pour spécifier la source des données d'entrée lorsque vous exécuterez la tâche de traitement à l'étape 4.
with open('manifest.json', 'w') as fp:
json.dump(manifest, fp)
s3.meta.client.upload_file('manifest.json', s3_bucket, s3_folder + '/' + 'manifest.json')
os.remove('manifest.json')
s3_manifest_uri = f's3://{s3_bucket}/{s3_folder}/manifest.json'Maintenant que vous avez créé les fichiers manifestes d'entrée et que vous les avez téléchargés, vous pouvez écrire un script qui traite vos données dans le cadre de la tâche de traitement. Il traite les données des images satellites, calcule le NDVI, puis renvoie les résultats vers un autre emplacement Amazon S3.
Écrire un script qui calcule le NDVI
Amazon SageMaker Studio Classic prend en charge l'utilisation de la commande %%writefile cell magic. Après avoir exécuté une cellule avec cette commande, son contenu sera enregistré dans votre répertoire Studio Classic local. Il s'agit d'un code spécifique au calcul du NDVI. Toutefois, les éléments suivants peuvent être utiles lorsque vous écrivez votre propre script pour une tâche de traitement :
-
Dans votre conteneur de tâches de traitement, les chemins locaux à l'intérieur du conteneur doivent commencer par
/opt/ml/processing/. Dans cet exemple,input_data_path = '/opt/ml/processing/input_data/'etprocessed_data_path = '/opt/ml/processing/output_data/'sont spécifiés de cette manière. -
Avec Amazon SageMaker Processing, un script exécuté par une tâche de traitement peut télécharger vos données traitées directement sur Amazon S3. Pour ce faire, assurez-vous que le rôle d'exécution associé à votre
ScriptProcessorinstance possède les conditions requises pour accéder au compartiment S3. Vous pouvez également spécifier un paramètre de sortie lorsque vous exécutez votre tâche de traitement. Pour en savoir plus, consultez le fonctionnement de l'.run()APIdans le SDK Amazon SageMaker Python. Dans cet exemple de code, les résultats du traitement des données sont chargés directement sur Amazon S3. -
Pour gérer la taille de l'Amazon EBScontainer associé à votre tâche de traitement, utilisez le
volume_size_in_gbparamètre. La taille par défaut des conteneurs est de 30 Go. Vous pouvez également éventuellement utiliser la bibliothèque Python Garbage Collectorpour gérer le stockage dans votre conteneur Amazon EBS. L'exemple de code suivant charge les tableaux dans le conteneur de tâches de traitement. Lorsque les tableaux s'accumulent et remplissent la mémoire, la tâche de traitement se bloque. Pour éviter ce crash, l'exemple suivant contient des commandes qui suppriment les tableaux du conteneur de la tâche de traitement.
%%writefile compute_ndvi.py
import os
import pickle
import sys
import subprocess
import json
import rioxarray
if __name__ == "__main__":
print("Starting processing")
input_data_path = '/opt/ml/processing/input_data/'
input_files = []
for current_path, sub_dirs, files in os.walk(input_data_path):
for file in files:
if file.endswith(".json"):
input_files.append(os.path.join(current_path, file))
print("Received {} input_files: {}".format(len(input_files), input_files))
items = []
for input_file in input_files:
full_file_path = os.path.join(input_data_path, input_file)
print(full_file_path)
with open(full_file_path, 'r') as f:
items.append(json.load(f))
items = [item for sub_items in items for item in sub_items]
for item in items:
red_uri = item["Assets"]["red"]["Href"]
nir_uri = item["Assets"]["nir"]["Href"]
red = rioxarray.open_rasterio(red_uri, masked=True)
nir = rioxarray.open_rasterio(nir_uri, masked=True)
ndvi = (nir - red)/ (nir + red)
file_name = 'ndvi_' + item["Id"] + '.tif'
output_path = '/opt/ml/processing/output_data'
output_file_path = f"{output_path}/{file_name}"
ndvi.rio.to_raster(output_file_path)
print("Written output:", output_file_path)Vous disposez à présent d'un script capable de calculer le NDVI. Ensuite, vous pouvez créer une instance de la tâche de traitement ScriptProcessor et exécuter votre tâche de traitement.
Création d'une instance de la ScriptProcessor classe
Cette démo utilise la ScriptProcessor.run() méthode.
from sagemaker.processing import ScriptProcessor, ProcessingInput, ProcessingOutput
image_uri = '081189585635.dkr.ecr.us-west-2.amazonaws.com/sagemaker-geospatial-v1-0:latest'
processor = ScriptProcessor(
command=['python3'],
image_uri=image_uri,
role=execution_role_arn,
instance_count=4,
instance_type='ml.m5.4xlarge',
sagemaker_session=sm_session
)
print('Starting processing job.')Lorsque vous démarrez votre tâche de traitement, vous devez spécifier un ProcessingInput
-
Le chemin d'accès au fichier manifeste que vous avez créé à l'étape 2,
s3_manifest_uri. Il s'agit de la source des données d'entrée du conteneur. -
Le chemin vers lequel vous souhaitez que les données d'entrée soient enregistrées dans le conteneur. Il doit correspondre au chemin que vous avez indiqué dans votre script.
-
Utilisez le
s3_data_typeparamètre pour spécifier l'entrée sous la forme"ManifestFile".
s3_output_prefix_url = f"s3://{s3_bucket}/{s3_folder}/output"
processor.run(
code='compute_ndvi.py',
inputs=[
ProcessingInput(
source=s3_manifest_uri,
destination='/opt/ml/processing/input_data/',
s3_data_type="ManifestFile",
s3_data_distribution_type="ShardedByS3Key"
),
],
outputs=[
ProcessingOutput(
source='/opt/ml/processing/output_data/',
destination=s3_output_prefix_url,
s3_upload_mode="Continuous"
)
]
)L'exemple de code suivant utilise la .describe()méthode
preprocessing_job_descriptor = processor.jobs[-1].describe()
s3_output_uri = preprocessing_job_descriptor["ProcessingOutputConfig"]["Outputs"][0]["S3Output"]["S3Uri"]
print(s3_output_uri)Visualisez vos résultats à l'aide de matplotlib
Avec la bibliothèque Python Matplotlib.open_rasterio() API, puis calcule le NDVI à l'aide des bandes d'images nir et red issues du Sentinel-2 données satellitaires.
# Opens the python arrays
import rioxarray
red_uri = items[25]["Assets"]["red"]["Href"]
nir_uri = items[25]["Assets"]["nir"]["Href"]
red = rioxarray.open_rasterio(red_uri, masked=True)
nir = rioxarray.open_rasterio(nir_uri, masked=True)
# Calculates the NDVI
ndvi = (nir - red)/ (nir + red)
# Common plotting library in Python
import matplotlib.pyplot as plt
f, ax = plt.subplots(figsize=(18, 18))
ndvi.plot(cmap='viridis', ax=ax)
ax.set_title("NDVI for {}".format(items[25]["Id"]))
ax.set_axis_off()
plt.show()La sortie de l'exemple de code précédent est une image satellite sur laquelle sont superposées les valeurs NDVI. Une valeur NDVI proche de 1 indique la présence d'une grande quantité de végétation, et des valeurs proches de 0 indiquent qu'aucune végétation n'est présente.
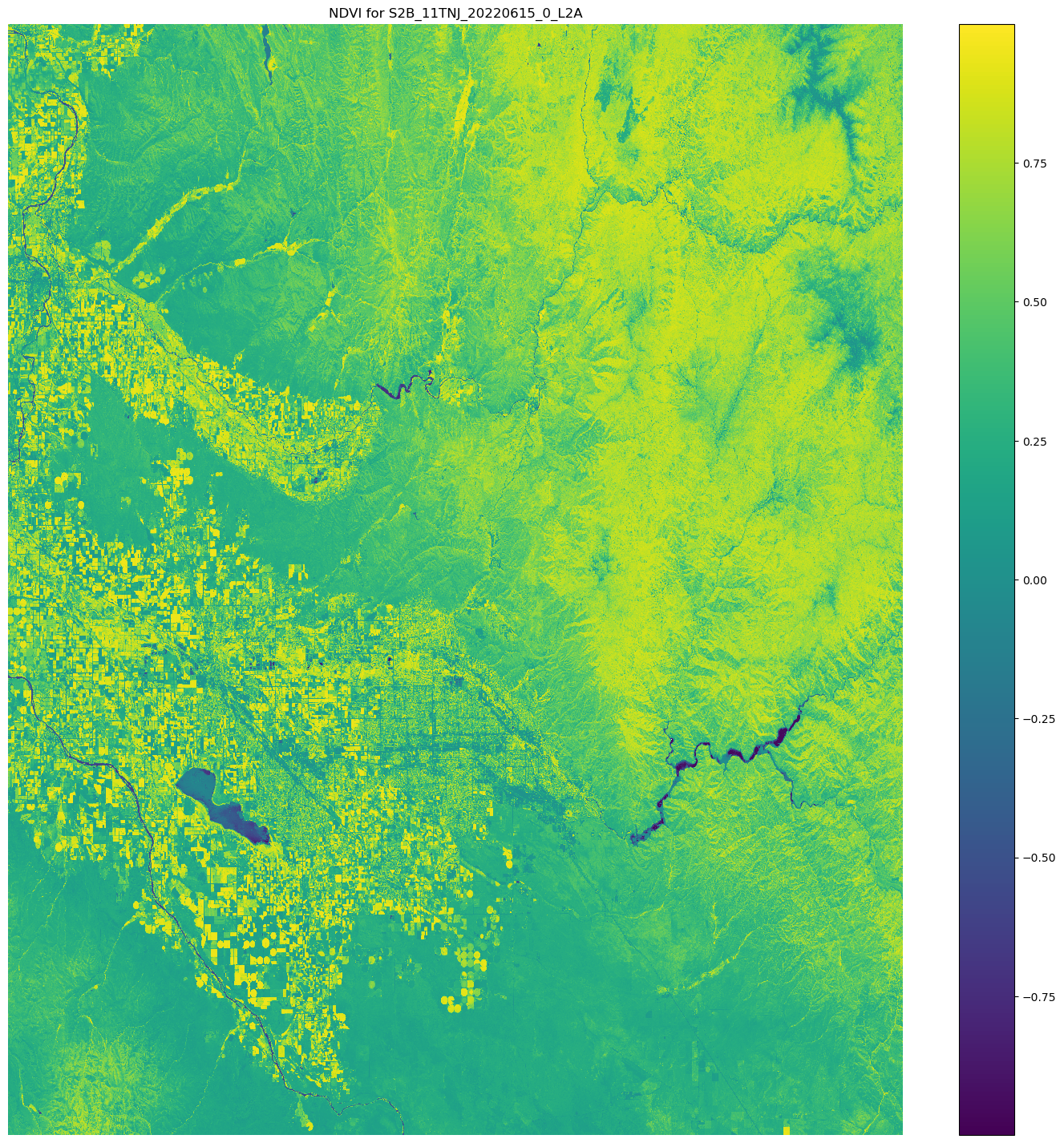
Ceci termine la démonstration d'utilisationScriptProcessor.