As traduções são geradas por tradução automática. Em caso de conflito entre o conteúdo da tradução e da versão original em inglês, a versão em inglês prevalecerá.
Usando ScriptProcessor para calcular o Índice de Vegetação por Diferença Normalizada (NDVI) usando Sentinel-2 dados de satélite
Os exemplos de código a seguir mostram como calcular o índice de vegetação de diferença normalizada de uma área geográfica específica usando a imagem geoespacial criada especificamente em um notebook Studio Classic e executar uma carga de trabalho em grande escala com o Amazon Processing SageMaker usando o SDK AI Python. ScriptProcessor
Essa demonstração também usa uma instância de notebook Amazon SageMaker Studio Classic que usa o kernel geoespacial e o tipo de instância. Para saber como criar uma instância de cadernos geoespaciais do Studio Classic, consulte Crie um notebook Amazon SageMaker Studio Classic usando a imagem geoespacial.
Você pode acompanhar essa demonstração em sua própria instância de caderno copiando e colando os seguintes trechos de código:
Consulte o Sentinel-2 coleta de dados raster usando SearchRasterDataCollection
Com search_raster_data_collection você pode consultar coleções de dados raster compatíveis. Este exemplo usa dados extraídos de Sentinel-2 satélites. A área de interesse (AreaOfInterest) especificada é a zona rural do norte de Iowa, e o intervalo de tempo (TimeRangeFilter) é de 1º de janeiro de 2022 a 30 de dezembro de 2022. Para ver as coleções de dados raster disponíveis em seu Região da AWS use list_raster_data_collections. Para ver um exemplo de código usando essa API, consulte ListRasterDataCollectionso Amazon SageMaker AI Developer Guide.
Nos exemplos de código a seguir, você usa o ARN associado a Sentinel-2 coleta de dados raster,arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8.
Uma solicitação de API search_raster_data_collection requer dois parâmetros:
-
Você precisa especificar um parâmetro
Arnque corresponda à coleção de dados raster que você deseja consultar. -
Você também precisa especificar um
RasterDataCollectionQueryparâmetro, que assume um Python dicionário.
O exemplo de código a seguir contém os pares de valores-chave necessários para o parâmetro RasterDataCollectionQuery salvo na variável search_rdc_query.
search_rdc_query = { "AreaOfInterest": { "AreaOfInterestGeometry": { "PolygonGeometry": { "Coordinates": [[ [ -94.50938680498298, 43.22487436936203 ], [ -94.50938680498298, 42.843474642037194 ], [ -93.86520004156142, 42.843474642037194 ], [ -93.86520004156142, 43.22487436936203 ], [ -94.50938680498298, 43.22487436936203 ] ]] } } }, "TimeRangeFilter": {"StartTime": "2022-01-01T00:00:00Z", "EndTime": "2022-12-30T23:59:59Z"} }
Para fazer a search_raster_data_collection solicitação, você deve especificar o ARN do Sentinel-2 coleta de dados raster:arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8. Você também precisa passar o dicionário Python que foi definido anteriormente, que especifica os parâmetros de consulta.
## Creates a SageMaker Geospatial client instance sm_geo_client= session.create_client(service_name="sagemaker-geospatial") search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query )
Os resultados dessa API não podem ser paginados. Para coletar todas as imagens de satélite retornadas pela operação search_raster_data_collection, você pode implementar um loop while. Isso é verificado para NextToken na resposta da API:
## Holds the list of API responses from search_raster_data_collection items_list = [] while search_rdc_response1.get('NextToken') and search_rdc_response1['NextToken'] != None: items_list.extend(search_rdc_response1['Items']) search_rdc_response1 = sm_geo_client.search_raster_data_collection( Arn='arn:aws:sagemaker-geospatial:us-west-2:378778860802:raster-data-collection/public/nmqj48dcu3g7ayw8', RasterDataCollectionQuery=search_rdc_query, NextToken=search_rdc_response1['NextToken'] )
A resposta da API retorna uma lista URLs abaixo da Assets chave correspondente a faixas de imagem específicas. Veja a seguir uma versão truncada da resposta da API. Algumas das faixas da imagem foram removidas para maior clareza.
{ 'Assets': { 'aot': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/AOT.tif' }, 'blue': { 'Href': 'https://sentinel-cogs.s3.us-west-2.amazonaws.com/sentinel-s2-l2a-cogs/15/T/UH/2022/12/S2A_15TUH_20221230_0_L2A/B02.tif' }, 'swir22-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/B12.jp2' }, 'visual-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/TCI.jp2' }, 'wvp-jp2': { 'Href': 's3://sentinel-s2-l2a/tiles/15/T/UH/2022/12/30/0/WVP.jp2' } }, 'DateTime': datetime.datetime(2022, 12, 30, 17, 21, 52, 469000, tzinfo = tzlocal()), 'Geometry': { 'Coordinates': [ [ [-95.46676936182894, 43.32623760511659], [-94.11293433656887, 43.347431265475954], [-94.09532154452742, 42.35884880571144], [-95.42776890002203, 42.3383710796791], [-95.46676936182894, 43.32623760511659] ] ], 'Type': 'Polygon' }, 'Id': 'S2A_15TUH_20221230_0_L2A', 'Properties': { 'EoCloudCover': 62.384969, 'Platform': 'sentinel-2a' } }
Na próxima seção, você cria um arquivo manifesto usando a chave 'Id' da resposta da API.
Crie um arquivo manifesto de entrada usando a chave Id da resposta da API search_raster_data_collection
Ao executar um trabalho de processamento, você deve especificar uma entrada de dados do Amazon S3. O tipo de dados de entrada pode ser um arquivo manifesto, que então aponta para os arquivos de dados individuais. Você também pode adicionar um prefixo a cada arquivo que você deseja processar. O exemplo de código a seguir define a pasta na qual seus arquivos manifesto serão gerados.
Use o SDK for Python (Boto3) para obter o bucket padrão e o ARN da função de execução associada à sua instância de caderno Studio:
sm_session = sagemaker.session.Session() s3 = boto3.resource('s3') # Gets the default excution role associated with the notebook execution_role_arn = sagemaker.get_execution_role() # Gets the default bucket associated with the notebook s3_bucket = sm_session.default_bucket() # Can be replaced with any name s3_folder ="script-processor-input-manifest"
Em seguida, você cria um arquivo manifesto. Ele conterá as imagens URLs de satélite que você deseja processar ao executar seu trabalho de processamento posteriormente na etapa 4.
# Format of a manifest file manifest_prefix = {} manifest_prefix['prefix'] = 's3://' + s3_bucket + '/' + s3_folder + '/' manifest = [manifest_prefix] print(manifest)
O exemplo de código a seguir retorna o URI do S3 em que seus arquivos manifesto serão criados.
[{'prefix': 's3://sagemaker-us-west-2-111122223333/script-processor-input-manifest/'}]
Todos os elementos de resposta da resposta search_raster_data_collection não são necessários para executar a tarefa de processamento.
O trecho de código a seguir remove os elementos desnecessários 'Properties', 'Geometry' e 'DateTime'. O par de valores-chave 'Id', 'Id': 'S2A_15TUH_20221230_0_L2A', contém o ano e o mês. O exemplo de código a seguir analisa esses dados para criar novas chaves no Python dicionáriodict_month_items. Os valores são os ativos retornados da consulta SearchRasterDataCollection.
# For each response get the month and year, and then remove the metadata not related to the satelite images. dict_month_items = {} for item in items_list: # Example ID being split: 'S2A_15TUH_20221230_0_L2A' yyyymm = item['Id'].split("_")[2][:6] if yyyymm not in dict_month_items: dict_month_items[yyyymm] = [] # Removes uneeded metadata elements for this demo item.pop('Properties', None) item.pop('Geometry', None) item.pop('DateTime', None) # Appends the response from search_raster_data_collection to newly created key above dict_month_items[yyyymm].append(item)
Este exemplo de código carrega o dict_month_items para o Amazon S3 como um objeto JSON usando a operação de API .upload_file()
## key_ is the yyyymm timestamp formatted above ## value_ is the reference to all the satellite images collected via our searchRDC query for key_, value_ in dict_month_items.items(): filename = f'manifest_{key_}.json' with open(filename, 'w') as fp: json.dump(value_, fp) s3.meta.client.upload_file(filename, s3_bucket, s3_folder + '/' + filename) manifest.append(filename) os.remove(filename)
Esse exemplo de código carrega um arquivo principal manifest.json que aponta para todos os outros manifestos enviados para o Amazon S3. Também salva o caminho para uma variável local: s3_manifest_uri. Você usará essa variável novamente para especificar a origem dos dados de entrada ao executar o trabalho de processamento na etapa 4.
with open('manifest.json', 'w') as fp: json.dump(manifest, fp) s3.meta.client.upload_file('manifest.json', s3_bucket, s3_folder + '/' + 'manifest.json') os.remove('manifest.json') s3_manifest_uri = f's3://{s3_bucket}/{s3_folder}/manifest.json'
Agora que você criou os arquivos manifesto de entrada e os carregou, você pode escrever um script que processe seus dados na tarefa de processamento. Ele processa os dados das imagens de satélite, calcula o NDVI e, em seguida, retorna os resultados para um local diferente do Amazon S3.
Escreva um script que calcule o NDVI
O Amazon SageMaker Studio Classic suporta o uso do comando %%writefile cell magic. Depois de executar uma célula com esse comando, seu conteúdo será salvo no diretório local do Studio Classic. Esse é um código específico para calcular o NDVI. No entanto, o seguinte pode ser útil quando você escreve seu próprio script para uma tarefa de processamento:
-
Em seu contêiner de trabalho de processamento, os caminhos locais dentro do contêiner devem começar com
/opt/ml/processing/. Neste exemplo,input_data_path = '/opt/ml/processing/input_data/'eprocessed_data_path = '/opt/ml/processing/output_data/'são especificados dessa forma. -
Com o Amazon SageMaker Processing, um script executado por uma tarefa de processamento pode carregar seus dados processados diretamente para o Amazon S3. Para fazer isso, certifique-se de que o perfil de execução associada à sua instância
ScriptProcessortenha os requisitos necessários para acessar o bucket do S3. Você também pode especificar um parâmetro de saídas ao executar seu trabalho de processamento. Para saber mais, consulte a operação da.run()APIno SDK do Amazon SageMaker Python. Neste exemplo de código, os resultados do processamento de dados são carregados diretamente para o Amazon S3. -
Para gerenciar o tamanho da Amazon EBScontainer anexada ao seu processamento, use o
volume_size_in_gbparâmetro. O tamanho padrão dos contêineres é 30 GB. Opcionalmente, você também pode usar Coleta de resíduosda biblioteca Python para gerenciar o armazenamento em seu contêiner do Amazon EBS. O exemplo de código a seguir carrega as matrizes no contêiner do trabalho de processamento. Quando as matrizes se acumulam e preenchem a memória, a tarefa de processamento falha. Para evitar essa falha, o exemplo a seguir contém comandos que removem as matrizes do contêiner do trabalho de processamento.
%%writefile compute_ndvi.py import os import pickle import sys import subprocess import json import rioxarray if __name__ == "__main__": print("Starting processing") input_data_path = '/opt/ml/processing/input_data/' input_files = [] for current_path, sub_dirs, files in os.walk(input_data_path): for file in files: if file.endswith(".json"): input_files.append(os.path.join(current_path, file)) print("Received {} input_files: {}".format(len(input_files), input_files)) items = [] for input_file in input_files: full_file_path = os.path.join(input_data_path, input_file) print(full_file_path) with open(full_file_path, 'r') as f: items.append(json.load(f)) items = [item for sub_items in items for item in sub_items] for item in items: red_uri = item["Assets"]["red"]["Href"] nir_uri = item["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) ndvi = (nir - red)/ (nir + red) file_name = 'ndvi_' + item["Id"] + '.tif' output_path = '/opt/ml/processing/output_data' output_file_path = f"{output_path}/{file_name}" ndvi.rio.to_raster(output_file_path) print("Written output:", output_file_path)
Agora você tem um script que pode calcular o NDVI. Em seguida, você pode criar uma instância do ScriptProcessor e executar sua tarefa de processamento.
Criando uma instância da classe ScriptProcessor
Esta demonstração usa a ScriptProcessor.run().
from sagemaker.processing import ScriptProcessor, ProcessingInput, ProcessingOutput image_uri ='081189585635.dkr.ecr.us-west-2.amazonaws.com/sagemaker-geospatial-v1-0:latest'processor = ScriptProcessor( command=['python3'], image_uri=image_uri, role=execution_role_arn, instance_count=4, instance_type='ml.m5.4xlarge', sagemaker_session=sm_session ) print('Starting processing job.')
Ao iniciar seu trabalho de processamento, você precisa especificar um objeto ProcessingInput
-
O caminho para o arquivo manifesto que você criou na etapa 2,
s3_manifest_uri. Essa é a fonte dos dados de entrada para o contêiner. -
O caminho para onde você deseja que os dados de entrada sejam salvos no contêiner. Isso deve corresponder ao caminho que você especificou em seu script.
-
Use o parâmetro
s3_data_typepara especificar a entrada como"ManifestFile".
s3_output_prefix_url = f"s3://{s3_bucket}/{s3_folder}/output" processor.run( code='compute_ndvi.py', inputs=[ ProcessingInput( source=s3_manifest_uri, destination='/opt/ml/processing/input_data/', s3_data_type="ManifestFile", s3_data_distribution_type="ShardedByS3Key" ), ], outputs=[ ProcessingOutput( source='/opt/ml/processing/output_data/', destination=s3_output_prefix_url, s3_upload_mode="Continuous" ) ] )
O exemplo de código a seguir usa o método .describe()
preprocessing_job_descriptor = processor.jobs[-1].describe() s3_output_uri = preprocessing_job_descriptor["ProcessingOutputConfig"]["Outputs"][0]["S3Output"]["S3Uri"] print(s3_output_uri)
Visualizando seus resultados usando matplotlib
Com a biblioteca Matplotlib.open_rasterio() API e, em seguida, calcula o NDVI usando as bandas de red imagem nir e do Sentinel-2 dados de satélite.
# Opens the python arrays import rioxarray red_uri = items[25]["Assets"]["red"]["Href"] nir_uri = items[25]["Assets"]["nir"]["Href"] red = rioxarray.open_rasterio(red_uri, masked=True) nir = rioxarray.open_rasterio(nir_uri, masked=True) # Calculates the NDVI ndvi = (nir - red)/ (nir + red) # Common plotting library in Python import matplotlib.pyplot as plt f, ax = plt.subplots(figsize=(18, 18)) ndvi.plot(cmap='viridis', ax=ax) ax.set_title("NDVI for {}".format(items[25]["Id"])) ax.set_axis_off() plt.show()
A saída do exemplo de código anterior é uma imagem de satélite com os valores de NDVI sobrepostos nela. Um valor de NDVI próximo a 1 indica que muita vegetação está presente e valores próximos a 0 indicam que nenhuma vegetação está presente.
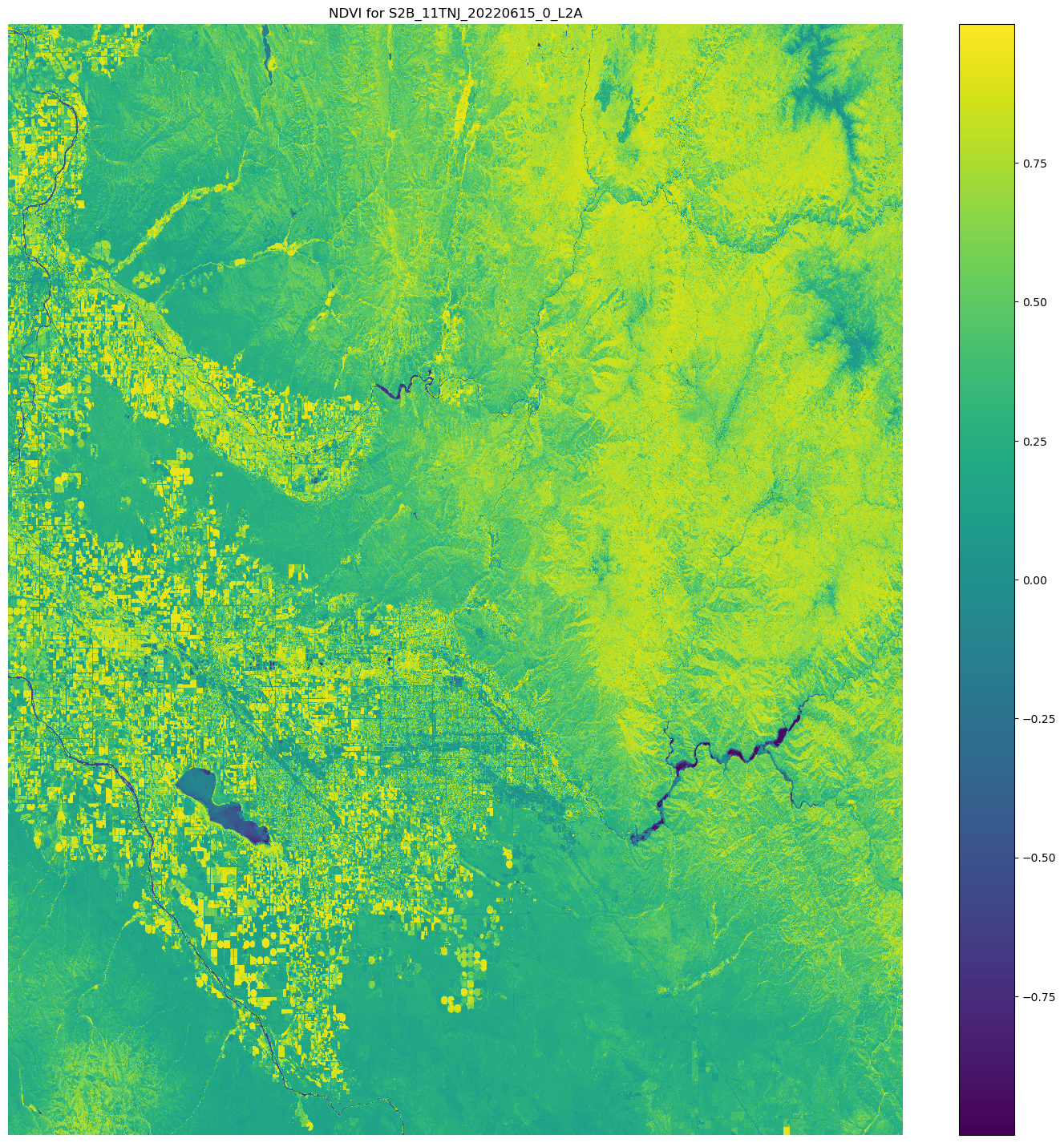
Isso conclui a demonstração do uso de ScriptProcessor.